模仿
MD最好的入门方式就是模仿!!!!!
找一篇难度适中的,软件,研究点都比较贴近自己方向的论文进行模仿。作为初学的我很幸运找到了这篇论文,在模仿过程中也逐渐的掌握了lammps的相关命令,如果大家也觉得适合的话,也可以进行模仿练习。比较贴近的意思是眼光要广,只要那篇论文有涉及到你的研究方向,哪怕是一个参数,你都值得一看。
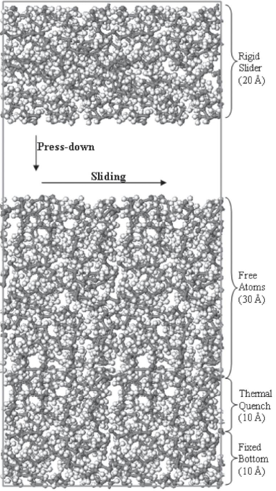
Dai L, Minn M, Satyanarayana N, Sinha SK, Tan VBC. Identifying the mechanisms of polymer friction through molecular dynamics simulation. Langmuir 2011;27(24):14861–7. https://doi.org/10.1021/la202763r .
模型方面就是简单的聚合物块,但是具有以下几个特点:
- 多层结构,端面很平
- 密度为指定值
此次博客只对MS导出的多层结构进行结构优化至论文的构型
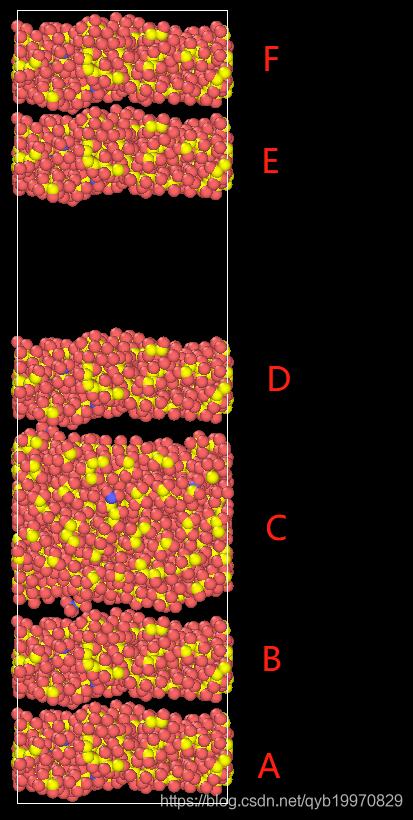
我分了ABCDEF六层,纯属为了方便起见,只有两个厚度的polymer layer,但是不影响后续的模仿学习,从这里就可以看出MS导出的模型完全达不到论文的初始模子,端面不齐,密度无法保证,总之就是很糟糕的模型。。我在MS也倒腾了很多次,依旧达不到想要的效果,所以需要在lammps中进行优化结构。
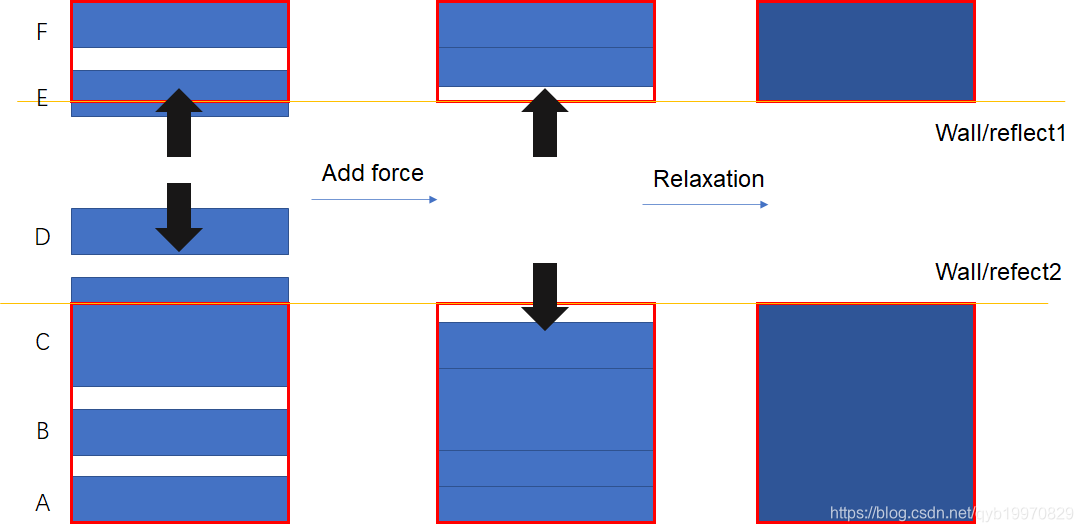
密度控制
所谓密度控制就是,让一定量的原子达到一定量的体积,所以我进行了一系列的压缩处理,用到的主要命令就是wall/reflect,
基本分为以下三步:
- 确定目标密度下聚合物块的高度
- add force使ABCD和EF压缩到指定高度
- 弛豫到wall/reflect边界处
部分in文件如下:
#-----------------------初始模拟系统设置
dimension 3
units real
atom_style full
boundary p p f#-----------------------近邻设置
neighbor 1.0 bin
neigh_modify delay 0 every 10 check yes#-----------------------coul/long预设置
kspace_style ewald 1.0e-4 #用于周期性边界
kspace_modify slab 3.0 #可用于ppf边界#-----------------------力场设置
pair_style lj/class2/coul/long 10.0
bond_style class2
angle_style class2
dihedral_style class2
improper_style class2#-----------------------原子坐标读入
read_data ABCDEF.data#-----------------------结构参数(CH原子数量)
variable C_number_A equal 400 #A层碳原子数目
variable C_number_B equal 400 #B层碳原子数目
variable C_number_C equal 900 #C层碳原子数目
variable C_number_D equal 400 #D层碳原子数目
variable C_number_E equal 400 #E层碳原子数目
variable C_number_F equal 400 #F层碳原子数目variable H_number_A equal 808 #A层氢原子数目
variable H_number_B equal 808 #B层氢原子数目
variable H_number_C equal 1818 #C层氢原子数目
variable H_number_D equal 808 #D层氢原子数目
variable H_number_E equal 808 #E层氢原子数目
variable H_number_F equal 808 #F层氢原子数目variable C_number_01 equal ${
C_number_A}+${
C_number_B}+${
C_number_C}+${
C_number_D} #ABCD层C原子数量
variable C_number_02 equal ${
C_number_E}+${
C_number_F} #EF层C原子数量
variable H_number_01 equal ${
H_number_A}+${
H_number_B}+${
H_number_C}+${
H_number_D} #ABCD层H原子数量
variable H_number_02 equal ${
H_number_E}+${
H_number_F} #EF层H原子数量variable Atom_number_ABCD equal ${
C_number_01}+${
H_number_01} #ABCD层原子数量
variable Atom_number_EF equal ${
C_number_02}+${
H_number_02} #EF层原子数量#-----------------------密度参数设定
variable target_density equal 0.94 #g/cm^3
variable C_mass equal 1.993e-23 #g
variable H_mass equal 1.674e-24 #gvariable target_mass_01 equal (${
C_number_01}*${
C_mass})+(${
H_number_01}*${
H_mass}) #ABCD层原子质量(g)
variable target_volume_01 equal ${
target_mass_01}/${
target_density} #ABCD层体积(cm^3)
variable target_height_01 equal (${
target_volume_01}*1e24)/(lx*ly) #ABCD层指定密度下厚度(埃)
variable reflect2 equal zlo+${
target_height_01} #指定密度下ABCD的z轴最高值#-----------------------力学参数设定
variable target_pressure equal 1e9 #目标压力设定,比例系数*1Gpa(pa)
variable total_force_E_F equal ${
target_pressure}*lx*ly*1e-20 #EF层所受的总力(N)
variable atom_force_E_F equal ${
total_force_E_F}/(${
C_number_02}+${
H_number_02}) #EF层每一个原子受到的力(N)
variable f_coefficient equal 6.950166e-11
variable f_to_addforce equal ${
atom_force_E_F}/${
f_coefficient}
补充
- 为什么我用ppf,而不用pps?
聚合物采用的pcff力场,pair_style为lj/class2/coul/long, 需要额外增加kspace命令,恶心的是,这命令还只能用于周期性边界??!!!还好有kspace_modify,可以勉强用在ppf中,用f边界注意原子丢失情况,所以在非周期性边界中要活用wall/reflect命令!!! - 我的ABCD和EF之间隔得太远了,你们在模仿的时候看着来,大于截断距离就ok了。
- 进行压缩的时候最好用addforce,我一开始用的是move linear,会出现反向move的情况,这个在第二个gif中应该可以看到。还有一点,其实addforce也不是最好的,因为压缩的距离没法儿控制,这个需要人为加大步长,后续结合可视化软件看哪一步是最为合适结束addforce,然后弛豫到指定密度距离的。
- 如果是体系中只有一个集中的区域,例如体系只有ABCD或者EF,果断addforce,thermo_style的时候导出density看密度就ok,密度超了记住步数,跑那个步数就ok。
- 在最后导出data和restart文件
restart可以保证先前的group分类,这点很重要!!!!具体看manual。
#-----------------------创建data/restart文件
write_restart restart.optimization
write_data data.ABCDEF_OPTIMIZATION
模型优化效果图
ABCD压缩
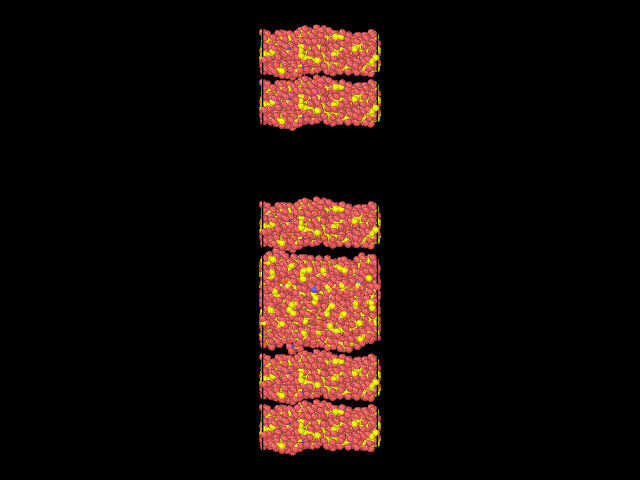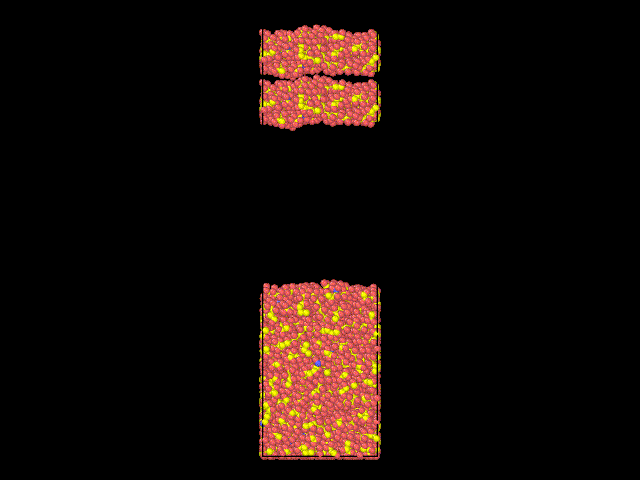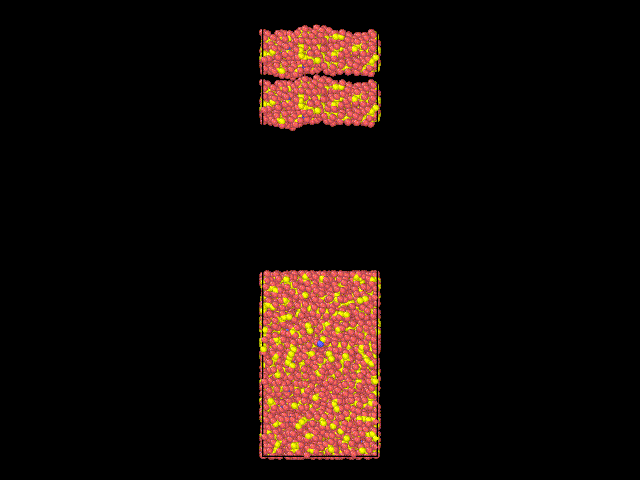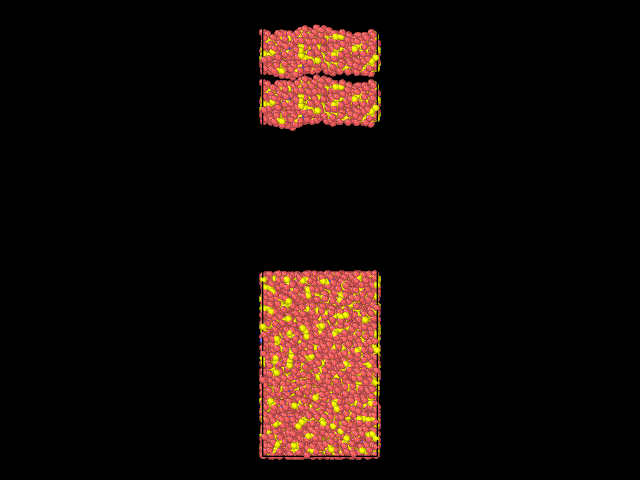
EF压缩
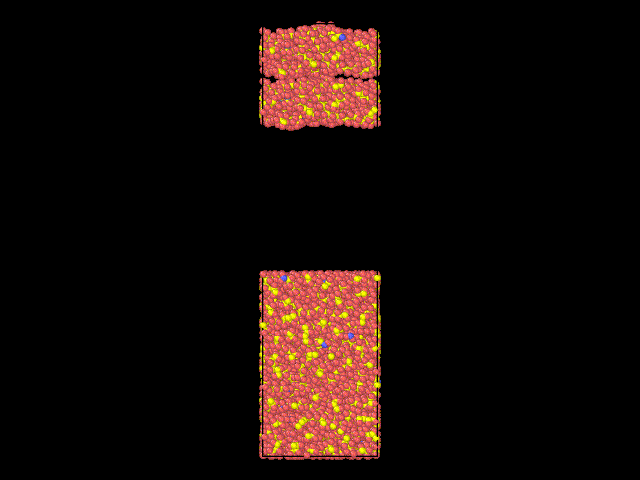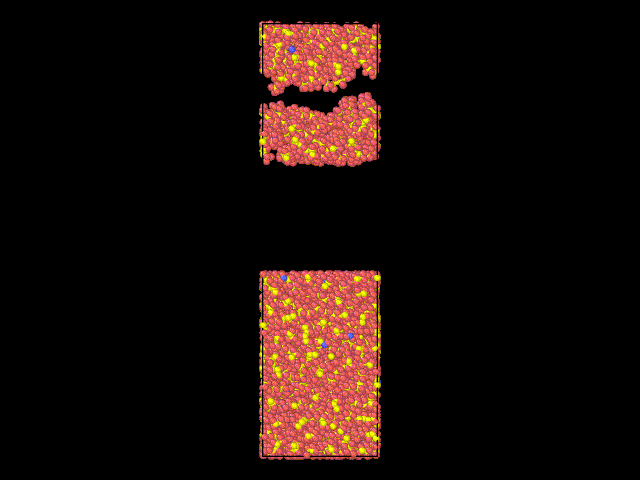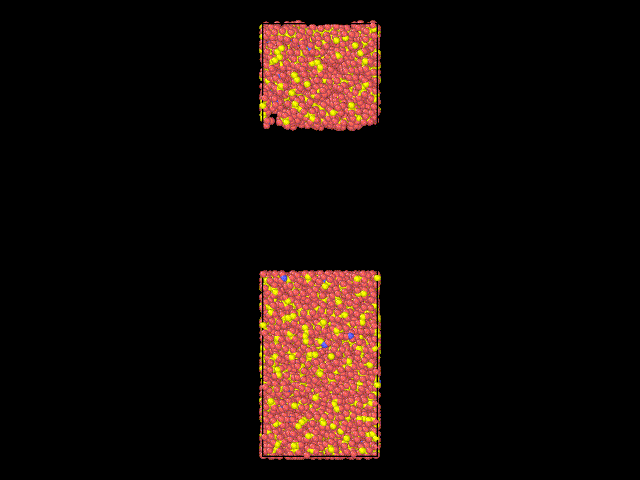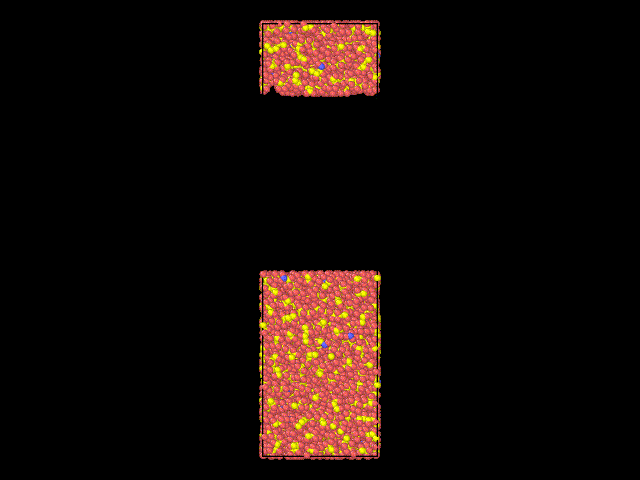
一个还算ok 的模型就出来了
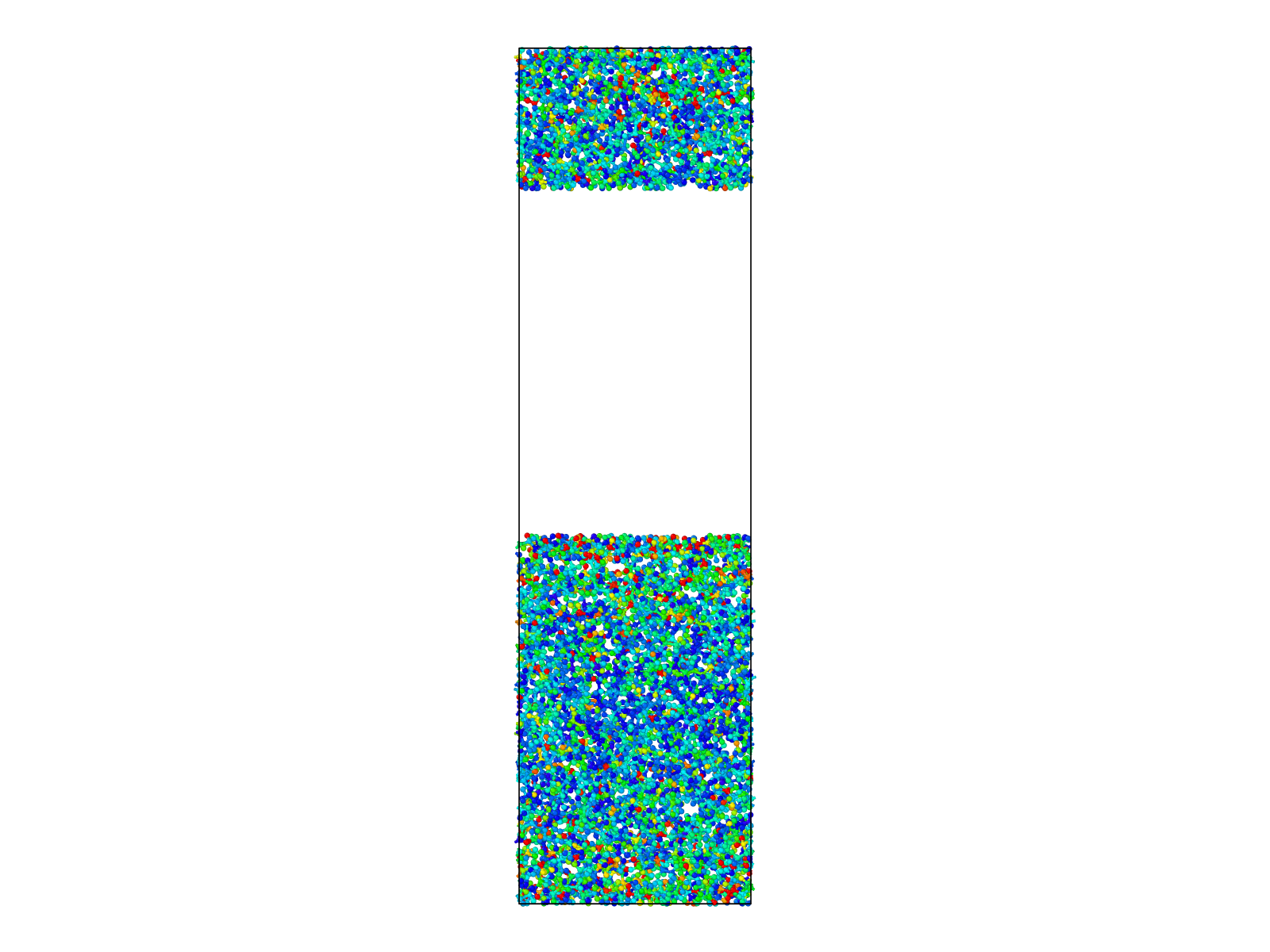
后续会继续模更新论文中的压力加载过程。。。。
参考文献
[1] Zhan S, Xu H, Duan H, Pan L, Jia D, Tu J et al. Molecular dynamics simulation of microscopic friction mechanisms of amorphous polyethylene. Soft Matter 2019;15(43):8827–39. https://doi.org/10.1039/c9sm01533g .
[2] Tong R-t, Han B, Quan Z-f, Liu G. Molecular dynamics simulation of friction and heat properties of Nano-texture GOLD film in space environment. Surface and Coatings Technology 2019;358:775–84. https://doi.org/10.1016/j.surfcoat.2018.11.084 .
[3] Ewen JP, Gao H, Müser MH, Dini D. Shear heating, flow, and friction of confined molecular fluids at high pressure. Phys Chem Chem Phys 2019;21(10):5813–23. https://doi.org/10.1039/c8cp07436d .
[4] Chen K, Wang L, Chen Y, Wang Q. Molecular dynamics simulation of microstructure evolution and heat dissipation of nanoscale friction. International Journal of Heat and Mass Transfer 2017;109:293–301. https://doi.org/10.1016/j.ijheatmasstransfer.2017.01.105 .
[5] Dai L, Sorkin V, Sha ZD, Pei QX, Branicio PS, Zhang YW. Molecular dynamics simulations on the frictional behavior of a perfluoropolyether film sandwiched between diamond-like-carbon coatings. Langmuir 2014;30(6):1573–9. https://doi.org/10.1021/la404680v .
[6] Uehara T. Molecular Dynamics Simulation of Stick-Slip Friction on a Metal Surface. AMM 2013;459:26–33. https://doi.org/10.4028/www.scientific.net/AMM.459.26 .
[7] Dong Y, Li Q, Martini A. Molecular dynamics simulation of atomic friction: A review and guide. Journal of Vacuum Science & Technology A: Vacuum, Surfaces, and Films 2013;31(3):30801. https://doi.org/10.1116/1.4794357 .
[8] Solar M, Meyer H, Gauthier C, Benzerara O, Schirrer R, Baschnagel J. Molecular dynamics simulations of the scratch test on linear amorphous polymer surfaces: A study of the local friction coefficient. Wear 2011;271(11-12):2751–8. https://doi.org/10.1016/j.wear.2011.05.026 .
[9] Dai L, Minn M, Satyanarayana N, Sinha SK, Tan VBC. Identifying the mechanisms of polymer friction through molecular dynamics simulation. Langmuir 2011;27(24):14861–7. https://doi.org/10.1021/la202763r .
[10] Sivebaek IM, Samoilov VN, Persson BNJ. Frictional properties of confined polymers. Eur Phys J E Soft Matter 2008;27(1):37–46. https://doi.org/10.1140/epje/i2008-10349-8 .
[11] Dias RA, Rapini M, Costa BV, Coura PZ. Temperature dependent molecular dynamic simulation of friction. Braz. J. Phys. 2006;36(3a):741–5. https://doi.org/10.1590/S0103-97332006000500029 .